

章节实例:第3章 碱基识别
Illumina平台碱基识别软件
(一) Alta-Cyclic
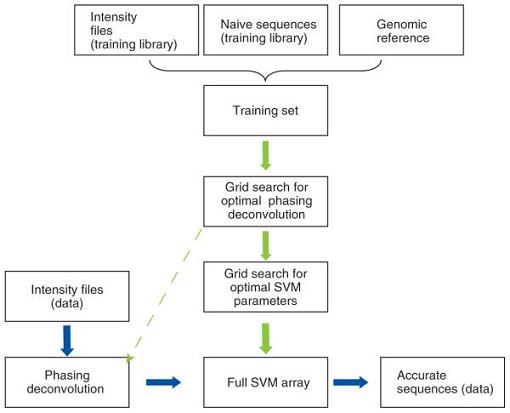
图3-1 Alta-Cyclic的处理流程(摘自Erlich et al., 2008)
图3-1显示了Alta-Cyclic的处理流程。Alta-Cyclic能够很好地将碱基识别长度延长至78个bp,并且相对较好地减少系统的偏差。由于统计的模型是使用了SVM,所以在运行Alta-Cyclic处理待处理的数据之前要进行一下数据集的训练过程,使得了Alta-Cyclic这款软件相对比较依赖训练集数据。Alta-Cyclic采用了三个来源的数据作为训练集,然后用贪婪算法寻找最优化的相重叠问题。此时加上强度质量文件,最优化SVM参数,从而得到SVM数据以及准确的序列文件。Alta-Cyclic的下载地址为:http://hannonlab.cshl.edu/Alta-Cyclic/main.html。
(二) Swift
Swift是使用C++语言编写的在Linux环境下可运行的软件,详细信息请参见:http://sgenomics.org/swift/ 。
Linux系统下可以通过访问该链接获得svn co:https://swiftng.svn.sourceforge.net/svnroot/swiftng/trunk。
利用Swift碱基识别的主要步骤概括如下:
1)矫正交叉干扰(crosstalk correction)
2)相位矫正(phasing correction)
3)纯化过滤(chastity filtering)
4)碱基识别(base calling)
经过上述步骤处理后就可以进行碱基识别的步骤了,这里所选择是最大强度的点。Swift的特点是可以输出一个点含有四个碱基概率的Fast4文件,此文件包含有每个点的存在的四个碱基的概率值,这样产生的有质量分数的文件在后续的匹配的过程中很有用,质量分数的值从Q6~Q30。
这一步骤的质量分数文件在后续出现的Base Calling软件中也被广泛的使用,用于对Base Calling过程作出评价。
(三) Ibis
Ibis的详细信息请参见:https://bioinf.eva.mpg.de/Ibis/,这里选择的版本为Ibis 1.1.6。
1)在Linux环境下Ibis的安装命令如下:
$ wget http://bioinf.eva.mpg.de/Ibis/ibis_1.1.6.tgz
2)在Linux下解压ibis_1.1.6.tgz的压缩文件:
$ tar vxzf ibis_1.1.6.tgz
$ cd Ibis_1.1.6
3)SVM构架
$ cd Ibis_SVMlight
$ make
$ cd
由于篇幅所限,其它能够处理Illumina平台数据的Base Calling软件这里不再叙述。
参考文献
Erlich, Y., et al. 2008. Alta-Cyclic: a self-optimizing base caller for next-generation sequencing. Nat Methods. 5(8): 679-682.